Comme le démontre la montée en puissance des inhibiteurs des checkpoints immunitaires, les progrès récents dans le domaine des thérapies antitumorales résultent d'une meilleure connaissance du microenvironnement tumoral (MET), avec, en particulier, l'étude des interactions entre les cellules immunitaires et tumorales. Ceci a profondément modifié notre manière d’appréhender l’étude des tumeurs et souligne l'importance d'intégrer pour les différents types cellulaires (cellules tumorales, cellules immunitaires, fibroblastes associés au cancer, cellules endothéliales, etc...) des informations sur leurs phénotypes, leurs fonctions et leurs localisations au sein de la tumeur afin d’évaluer leurs interactions physiques et fonctionnelles. Il est donc essentiel de pouvoir analyser un nombre important de paramètres biologiques simultanément (cluster de différenciation, cytokines, facteurs de transcription, etc...).
Dans ce but, la plateforme CIM s’est doté du premier système d'imagerie par cytométrie de masse, l’Hyperion. Développé par Fluidigm, l’Hyperion est un module d'imagerie qui, associé à l’Helios, un CyTOF de 3eme génération, permet l'analyse de cellules en suspension et tissulaire par spectrométrie de masse.
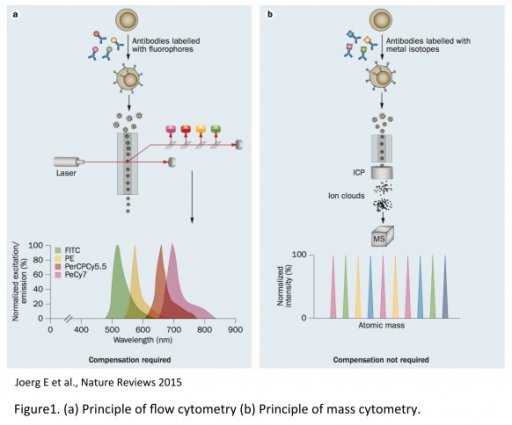
La cytométrie de masse utilise des anticorps ou sondes conjugués à des isotopes métalliques stables détectés par spectrométrie de masse (Time Of Flight, TOF). Cette technologie permet ainsi de surmonter les nombreuses limitations des technologies basées sur l’utilisation de la fluorescence telles que le chevauchement spectral, l'auto fluorescence, l'instabilité des fluorochromes ou le photo-blanchiment, qui rendent difficile la détection d’un spectre parmi l’ensemble des signaux optiques émis par l’échantillon.
En revanche, la spectrométrie de masse est capable de discriminer des isotopes de poids atomiques différents avec une grande précision. Cette différence fondamentale permet d'analyser simultanément beaucoup plus de paramètres cellulaires
(pour revue : Spitzer et al., Cell 2016).
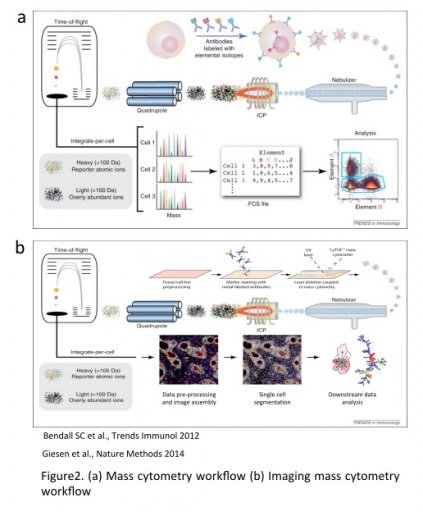
La cytométrie de masse supporte aujourd’hui un multiplexage élevé, jusqu'à 50 paramètres, pour 135 canaux disponibles sur l’Helios. Les isotopes métalliques stables peuvent être conjugués à des anticorps ou à des sondes afin de quantifier des protéines, des modifications post-traductionnelles, des produits de protéolyse, des activations de voies de signalisation, la transcription d'ARNm ou encore la synthèse d'ADN au sein d'une seule expérience. Les données générées peuvent alors être analysées et interprétées de la même manière que celles obtenues par FACs (fichiers FCS).
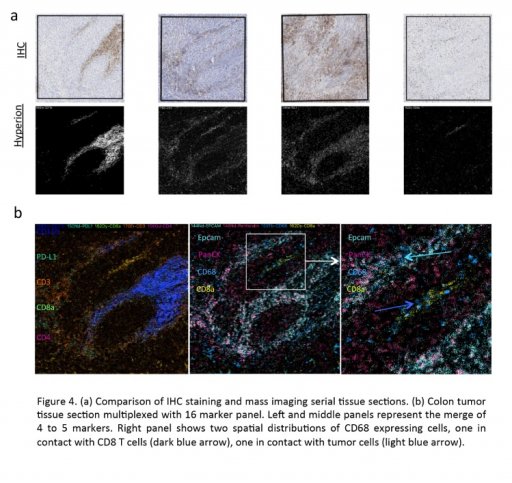
L'Hyperion permet l’analyse d’une coupe tissulaire préalablement marquée par un panel d‘anticorps ou de sondes. Après un marquage de type IHC ou HIS, un laser UV réalise une ablation à l’échelle subcellulaire du tissu (1µm2). Les événements produits sont ensuite analysés par l'Helios.
L’analyse de coupe tissulaire par Hyperion permet d'obtenir trois niveaux d'information :
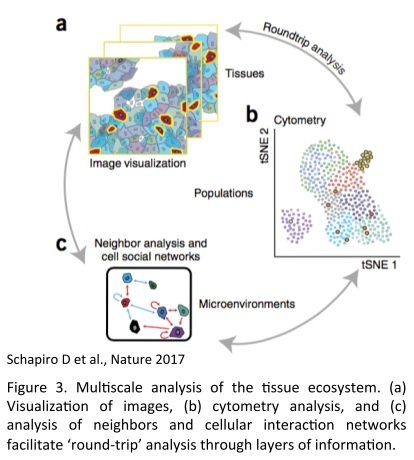
- L'identification phénotypique de cellules et de processus biologiques associés à leur distribution spatiale.
- Par une analyse non supervisée, la découverte de sous-types cellulaires au phénotype non-anticipé et l’étude de l'hétérogénéité de manière non biaisée.
- La caractérisation du réseau social de populations d’intérêt remis dans leur contexte environnemental.
Les applications de l'analyse cellulaire ou tissulaire basée sur la masse sont nombreuses et variées. Au-delà d'une étude approfondie et complète du microenvironnement tumoral , la découverte de biomarqueurs, l’étude de la pharmacocinétique de drogues dans la tumeur ou l'évaluation thérapeutique de combinaisons thérapeutiques est également possible. De plus, les données générées peuvent être comparées ou intégrées aux données générées avec des données " omiques " telles que génotype, transcriptome, protéome, matabolome ou imagerie.